Calidad de los datos de secuenciación
Resultados
*Basados en un control de calidad del secuenciador con tecnología DNBSEQ ™
En esta sección encontrarás una compilación de muchos de los datos generados por nuestro secuenciador con tecnología de DNA nanoballs. Entre ellos, se pueden observar gráficos y tablas sobre distribución de bases, la calidad de las lecturas y la eficiencia del array.
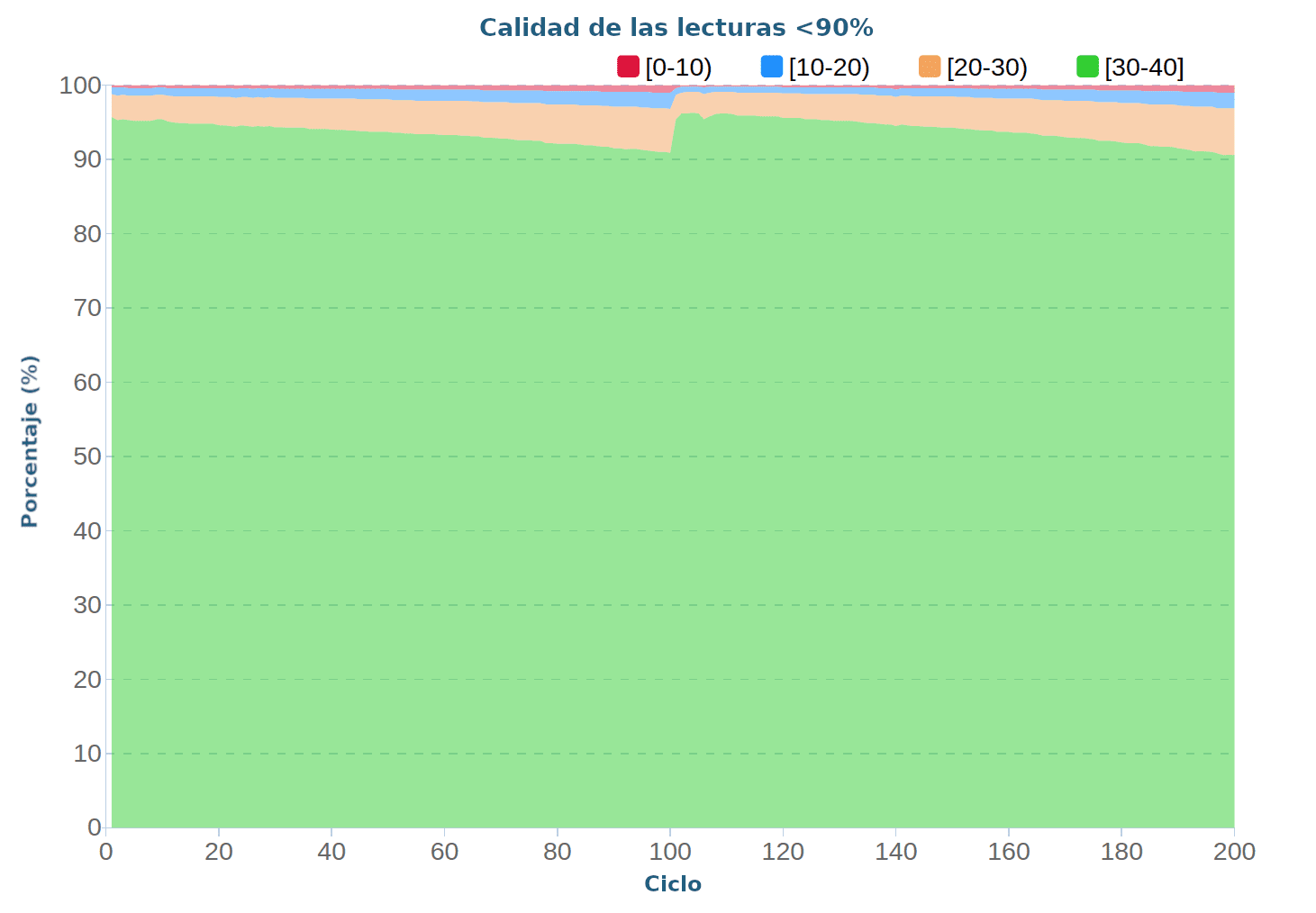
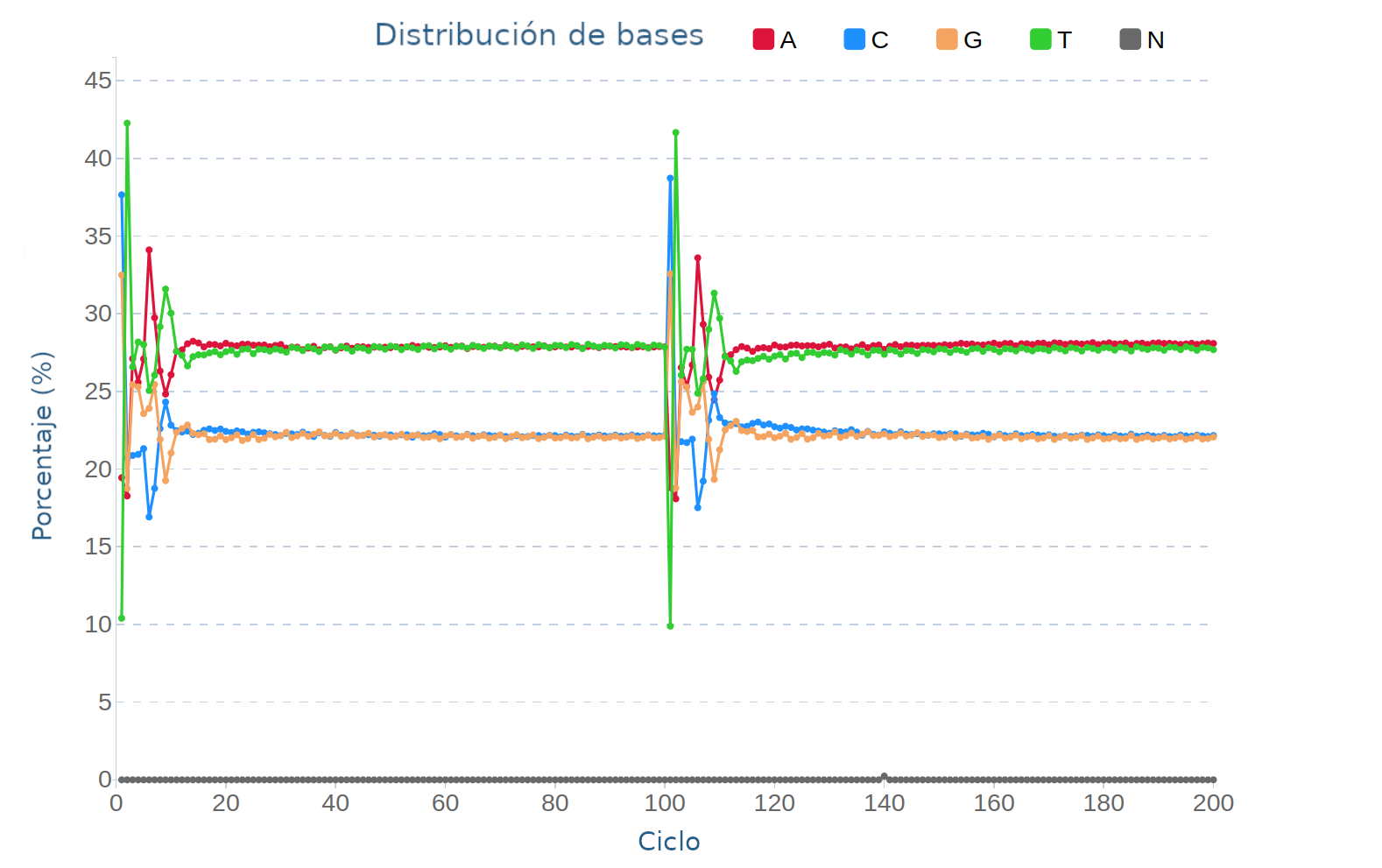
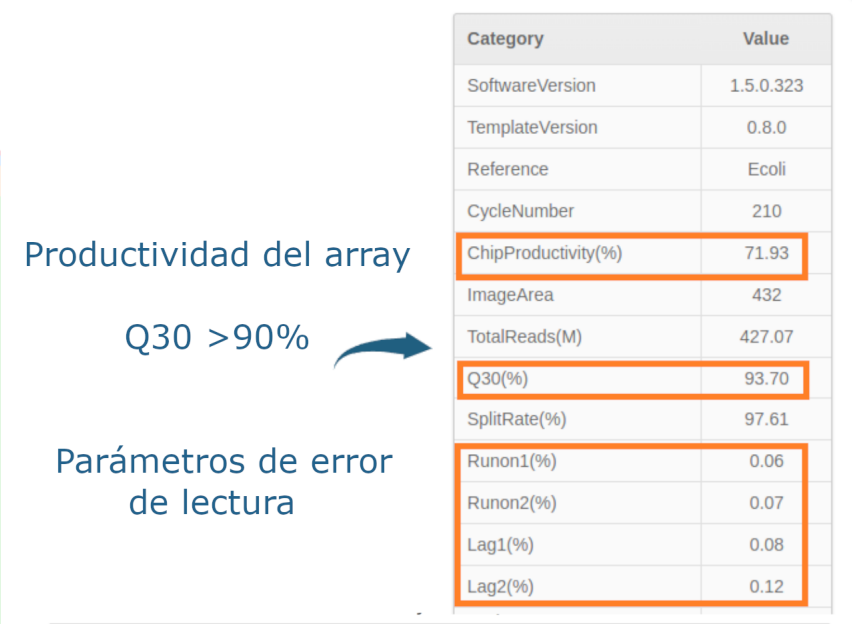
ESTUDIOS COMPARATIVOS DE LA TECNOLOGÍA DNBSEQ™
Rao, J. et al. evaluaron la eficacia para detectar variaciones en el número de copias (CNV) utilizando la secuenciación completa del genoma con la plataforma DNBSEQ™. En estudios previos, en los resultados, habían comprobado la eficacia de la plataforma para detectar variantes de nucleótido único (SNVs) e inserciones/ delecciones, este estudio se centró en la detección de CNVs. Los resultados indican que la capacidad de detección de CNVs en DNBSEQ™ es comparable a otras plataformas en términos de cantidad, longitud, distribución, sensibilidad y precisión en el genoma. Además, se destaca que las plataformas DNBSEQ™ ofrecen una visión más precisa de las CNVs de menor tamaño.
Rao, J., Peng, L., Liang, X. et al. (2020). Performance of copy number variants detection based on whole-genome sequencing by DNBSEQ platforms. BMC Bioinformatics 21, 518.
La ABRF realizó un estudio comparativo para evaluar la reproducibilidad, precisión y utilidad de varias plataformas de secuenciación NGS. Entre ellas se encontraban los instrumentos con tecnología DNBSEQ ™, que mostraron las tasas de error de secuenciación y de duplicados de lectura más bajas entre todos los instrumentos evaluados. Las muestras utilizadas fueron tanto de ADN humano como bacteriano, en las que dichos instrumentos DNBSEQ™ tuvieron una alta calidad de los datos y amplia cobertura. Además, el análisis de variantes reveló una mayor detección en cantidad de SNPs e INDELS con tasas de error muy bajas, siendo especialmente precisas en las regiones exómicas. También, tuvieron mayor precisión y sensibilidad, presentando así el menor número de falsos positivos y la mayor detección de inconsistencias mendelianas.
Pincha aquí para ver los resultados.
YFoox, J.et al. Performance assessment of DNA sequencing platforms in the ABRF Next-Generation Sequencing Study. Nat Biotechnol 39, 1129–1140 (2021).
¿Preparado para lograr tus metas de investigación?
Únete a los científicos, instituciones y facultativos que confían ya en OmicaLabs para una secuenciación NGS precisa y eficiente. Colaboremos juntos!
