Tecnología DNBSEQ™
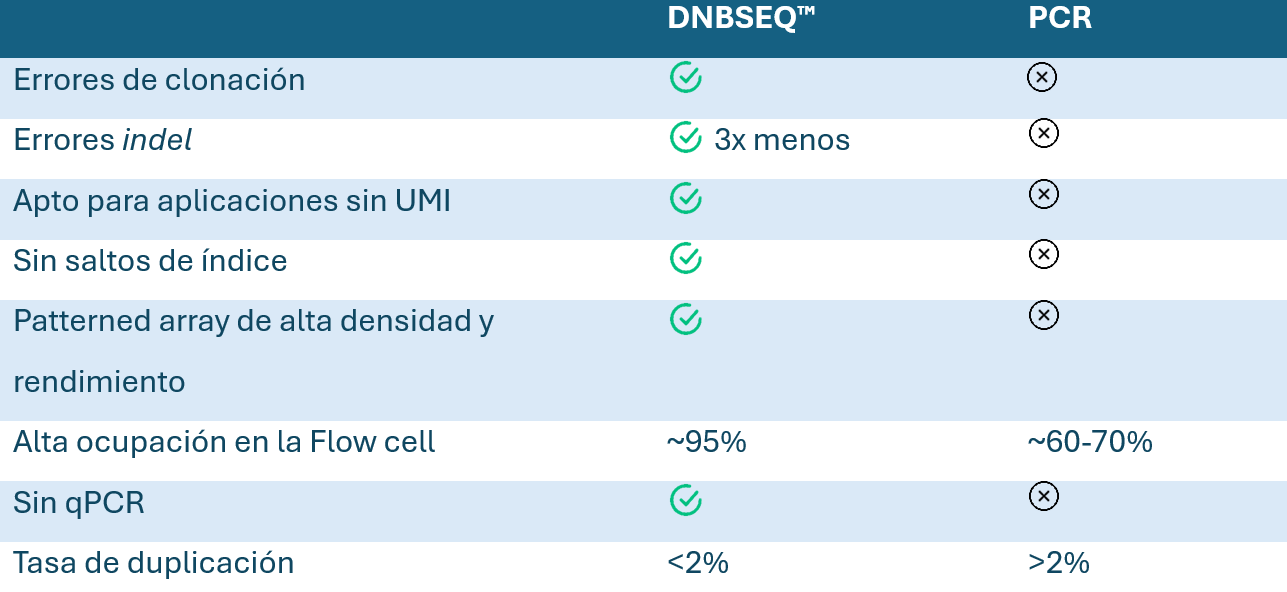
¿En qué se diferencia la tecnología DNBSEQ™ de otros métodos más tradicionales?
Mayor precisión — Más eficiencia
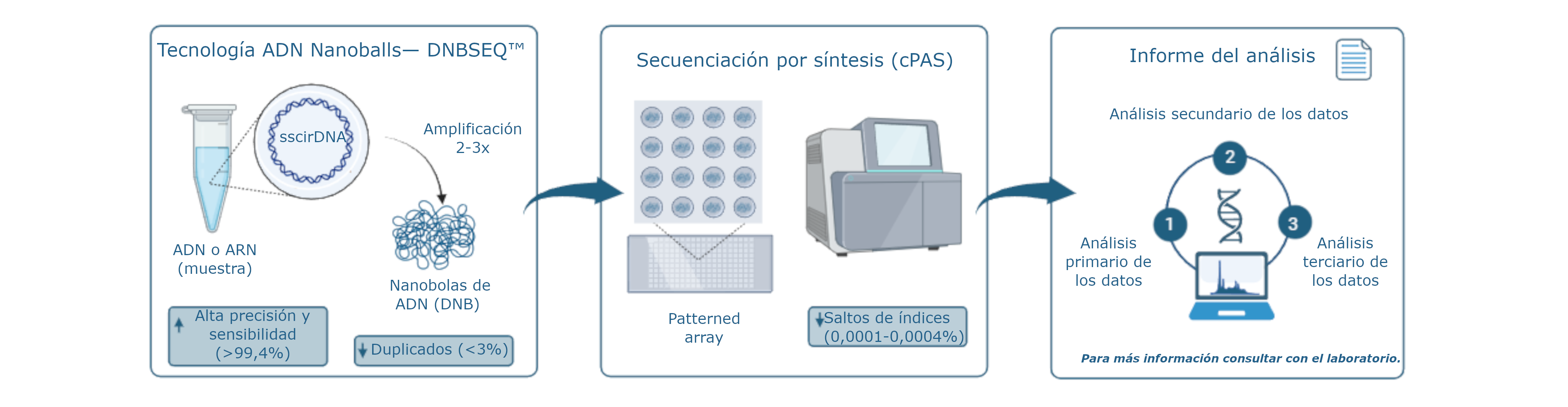
Funcionamiento de la tecnología DNBSEQ™ de las plataformas de secuenciación NGS
Para obtener más detalles sobre los datos y resultados de la secuenciación, haz clic aquí.
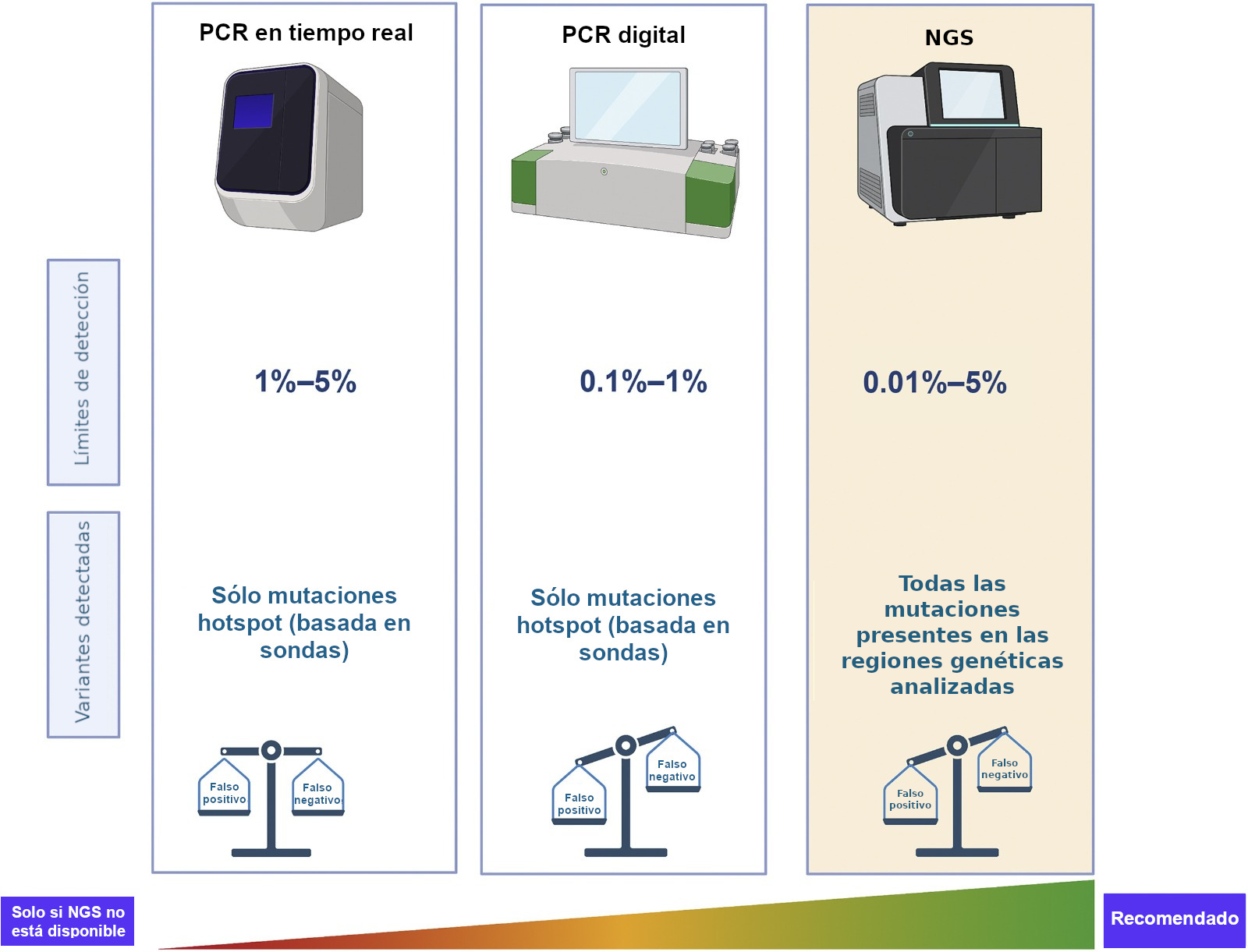
¿Cómo funciona esta tecnología de secuenciación?
Esta tecnología llamada DNBSEQ, se basa en nanobolas de ADN (DNB). Creadas durante la preparación de librerías, las DNB compactas se cargan en celdas de flujo y la secuencia se lee con cuatro sondas fluorescentes que reconocen cuatro bases de ADN. A continuación, se utilizan láseres para excitar las sondas, mientras se toman millones de imágenes para identificar las bases. Estas imágenes pasan por un análisis de imágenes y posteriormente un análisis bioinformático para identificar las variantes patogénicas y probablemente patogénicas encontradas.
Preparación de librerías
Durante la preparación de la librería de secuenciación, se crean las moléculas de ADN circular monocatenario (ssCirDNA). Los fragmentos típicos de ADN bicatenario, con secuencias adaptadoras en los extremos terminales, se calientan para generar ssADN. Un oligonucleótido se hibrida a ambos extremos del ssADN para formar círculos ‘nicked’ mientras que una ADN ligasa repara el ‘nick’ creando círculos monocatenarios completos.
Usando el círculo monocatenario se hace una replicación en círculo rodante (RCR) para crear miles de millones de nanobolas de ADN (DNB) en un solo tubo. Cada copia se realiza a partir del círculo de ADN original, lo que elimina los errores de amplificación clonal y reduce los sesgos de contenido GC y los abandonos que suele producir la PCR.
Secuenciación
Las DNBs se cargan en las ‘flow cells’, que tienen un array con un patrón de sitios de unión de ~200 nm uniformemente espaciados a distancias submicrométricas. Cada sitio se une a un único DNB, lo que garantiza un alto rendimiento de lecturas precisas con una señal nítida y sin interferencias de las demás DNBs adyacentes.
A partir de ahí, la química de síntesis combinatoria de sonda y anclaje (cPAS) hibrida los cebadores de secuenciación con los DNB y las sondas marcadas con fluorescencia y con terminación reversible son incorporadas por una ADN polimerasa patentada en ciclos de secuenciación consecutivos. A continuación, las sondas fluorescentes se excitan con luz láser y la matriz de DNB se visualiza mediante cámaras.